파이썬 프로파일링
26 Jul 2022파이썬 프로파일링
좋은 코드란 무엇일까? 좋은 코드에는 여러 가지 기준이 있지만 그 중 대표적인 것은 처리 속도와 메모리의 사용량일 것이다. 프로그램이 너무 느리거나 RAM을 많이 잡아먹는다면 문제가 되는 코드를 찾아내서 수정하고 싶을 것이다. 그럴 때 사용하기 좋은 것이 프로파일링이다.
프로파일링의 첫 번째 목표는 시스템의 어느 부분이 느린지, 어디서 RAM을 많이 쓰는지, 디스크 I/O나 네트워크 I/O를 과도하게 발생시키는 부분이 어디인지를 확인하는 것이다. 프로파일하면 보통 평소보다 10배에서 100배까지 실행 속도가 느려지는데, 최대한 실제 상황과 유사한 조건에서 테스트 하려면 테스트할 부분만 따로 떼어내서 테스트하자.
프로파일링의 전제로 실행 시간은 항상 약간의 편차가 있음을 기억하라. 코드의 실행 시간을 반복 측정하면 정규 분포가 나와야 한다. 그렇지 않다면 단순휘 무작위로 실행 시간이 달라지는 것을 보고 개선할 필요가 없는 코드를 개선할 수도 있다.
print와 데커레이터
처음 소개할 기본적인 프로파일링 기법은 time 패키지의 time.time(), 데커레이터를 활용한 시간 측정이다.
디버깅할 때와 마찬가지로 프로파일링에서도 print 문을 많이 사용한다. 금세 코드를 더럽히지만 잠깐 조사해야 할 때 유용하다.
import time
def solution():
...
start_time = time.time()
solution()
end_time = time.time()
secs = end_time - starttime
print(solution.__name__ + "함수 소요시간", secs, "초")
solution함수 소요시간 8.746789693832397 초
데커레이터는 print 문보다 조금 더 깔끔한 방법이다. 여기서는 시간을 측정하려는 함수 위에 코드를 한 줄 추가하면 된다.
import time
from functools import wraps
def timefn(fn):
@wraps(fn)
def measure_time(*args, **kwargs):
t1 = time.time()
result = fn(*args, **kwargs)
t2 = time.time()
print(f"@timefn: {fn.__name__} 함수 소요시간 {t2 - t1} 초 ")
return result
return measure_time
@timefn
def solution ():
...
@timefn: solution 함수 소요시간 8.808710813522339 초
위의 예제에서 timefn이라는 함수를 새로 정의한다. 내부에서 새로 정의한 measure_time 함수는 가변 길이 인자인 *args와 키워드 인자인 **kwargs를 받아, 실행하는 fn 함수로 넘겨준다. fn 함수를 호출하는 부분은 time.time()으로 감싸서 시간을 구하고, 함수의 이름과 함께 소요된 시간을 출력한다.
@wraps(fn)을 사용해서 데커레이터로 넘어온 함수 이름과 독스트링을 호출하는 함수에서 확인할 수 있도록 했다.
wraps 에 관해 이해하기 쉬운 내용을 담은 블로그 글이 있어서 링크를 남긴다.
https://velog.io/@doondoony/python-functools-wraps
cProfile 모듈 사용하기
cProfile은 표준 라이브러리에 내장된 프로파일링 도구로, C파이썬의 가상 머신 안에서 확인되는 모든 함수에 시간을 측정하는 장치를 연결한다. 이는 큰 오버헤드를 유발하지만 그만큼 더 많은 정보를 제공한다.
표준 라이브러리에서 제공하는 프로파일러는 cProfile과 profile이 있다. profile은 순수 파이썬 기반의 프로파일러로 cProfile보다 느리다. cProfile은 profile과 같은 인터페이스를 제공하며 오버헤드를 줄이려고 C로 작성했다.
cProfile을 사용하는 방법은 커맨드 라인에 명령어를 입력해주면 된다.
python -m cProfile -s cumulative s1.py
cProfile의 결과는 부모 함수에 따라 정렬되지 않으며 실행된 코드 블록 안의 모든 함수가 소비한 시간을 요약해 보여준다. 함수 안의 각 줄의 정보가 아닌 함수 호출 자체의 정보만 얻을 수 있다.
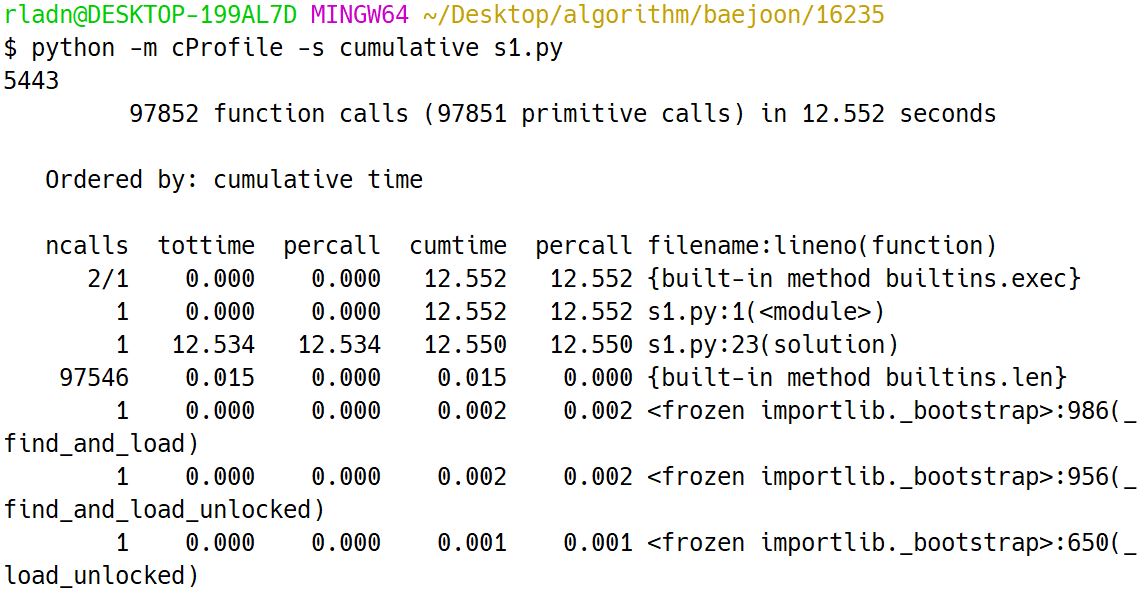
이전의 데커레이터와 timeit을 사용한 경우 8초 정도의 시간이 걸렸지만 cProfile을 사용한 위 예제의 경우 12초가 걸렸다. 이를 통해 함수가 소비한 시간을 추가로 알아내는 데 4초가량을 더 사용했다고 볼 수 있다.
커맨드 라인에 명령어를 입력해주는 방법 이외에 코드를 사용해 직접 cProfile을 사용할 수 있다.
from pstats import Stats
from cProfile import Profile
def solution():
...
if __name__ == "__main__":
profiler = Profile()
profiler.run('solution()')
stats = Stats(profiler)
stats.strip_dirs()
stats.sort_stats('cumulative')
stats.print_stats()
코드를 사용할 경우 실행 시간 4초가 소요되지 않는다.

line_profiler
line_profiler는 CPU 병목 원인을 찾아주는 강력한 도구다. line_profiler는 개별 함수를 한 줄씩 프로파일하므로, 먼저 cProfile을 사용해서 어떤 함수를 line_profiler로 자세히 살펴볼지 정하면 된다
line_profiler를 설치하려면 pip install line_profiler을 입력하면 된다.
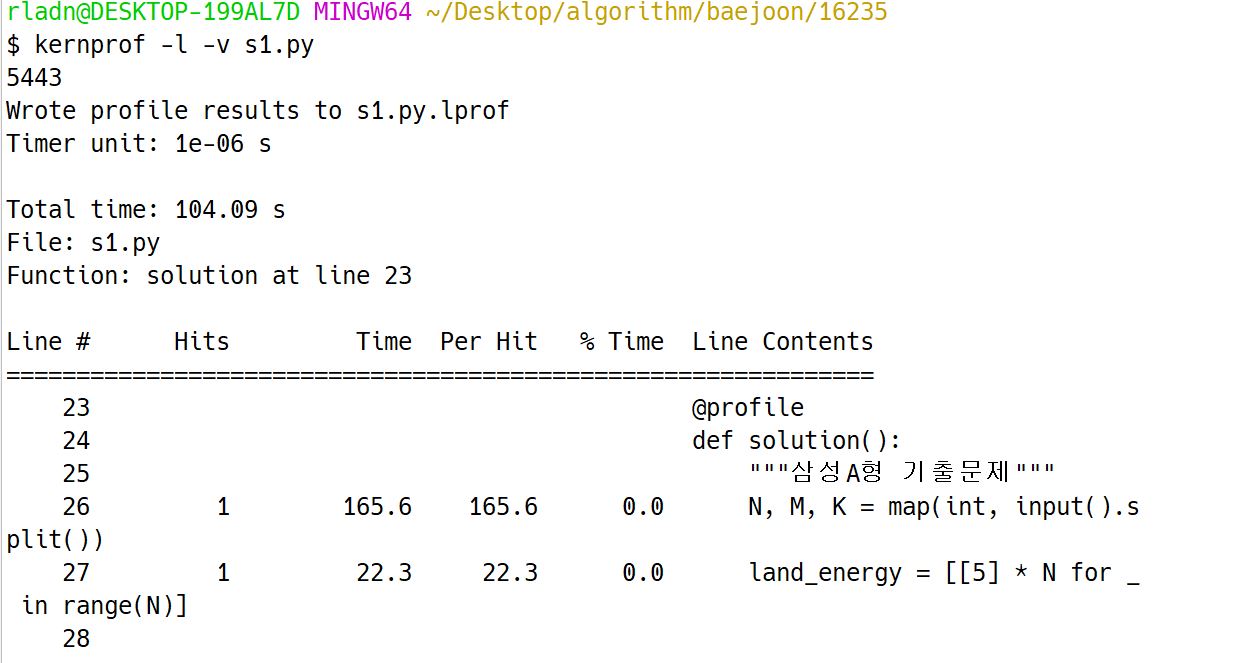
후에 @profile 데커레이터를 사용해 프로파일링 하고 싶은 함수를 선택한다. kernprof 스크립트는 코드를 실행하고 선택한 함수 각 줄의 CPU 시간 등 통계를 기록하는 데 사용한다.
-l 옵션은 함수 단위가 아니라 한 줄씩 프로파일하겠다는 옵션이며, -v 옵션은 출력 결과를 자세하게 보여준다.
@profile
def solution():
...
solution()
$ kernprof -l -v s1.py
timeit는 8초, cProfile은 12초가 걸렸지만 line_profile은 100초 가까운 시간이 걸린다..!
memory_profiler
line_profiler 패키지가 CPU 사용량을 측정하는 것처럼 memory_profiler는 메모리 사용량을 줄 단위로 측정해준다. memory_profiler는 line_profiler와 매우 흡사하게 작동하지만 훨씬 느리다. 보통 memory_profiler를 실행하면 실행속도가 평소보다 10배에서 100배까지 느려진다.
memory_profiler 는 pip install memory_profiler 명령으로 설치한 뒤, 커맨드 라인에 명령어를 입력한다.
python -m memory_profiler s1.py
같은 코드를 memory_profiler를 통해 프로파일링 한 경우 20분이 지나도 결과가 나오지 않아서 결과는 첨부하지 않겠다.
이러한 경우를 대비해서 코드를 일부분만 뗴어서 테스트하는 편이 좋다고 한다.
마치며
알고리즘을 풀다가 자신의 코드에 현자타임이 와서 프로파일링에 대한 글을 작성했다.
백준에 16235번 문제를 풀었고 실제 사용한 코드와 입력값은 아래와 같다.
from collections import defaultdict
import sys
sys.stdin = open("input.txt", "r")
input = sys.stdin.readline
def solution():
"""삼성A형 기출문제"""
N, M, K = map(int, input().split())
land_energy = [[5] * N for _ in range(N)]
A = [[] for _ in range(N)]
for i in range(N):
row = list(map(int, input().split()))
A[i] = row
total_tree = 0
trees = defaultdict(list)
for _ in range(M):
x, y, z = map(int, input().split())
trees[(x - 1, y - 1)].append(z)
total_tree += 1
delta = [(0, 1), (1, 0), (0, -1), (-1, 0), (-1, 1), (1, -1), (1, 1), (-1, -1)]
for _ in range(K):
grown = defaultdict(int)
for tree in trees:
len_tree = len(trees[tree])
for i in range(len_tree):
if land_energy[tree[0]][tree[1]] >= trees[tree][i]:
land_energy[tree[0]][tree[1]] -= trees[tree][i]
trees[tree][i] += 1
if trees[tree][i] % 5 == 0:
grown[tree] += 1
else:
for j in range(i, len_tree):
land_energy[tree[0]][tree[1]] += trees[tree][j] // 2
total_tree -= 1
trees[tree] = trees[tree][:i]
break
for tree in grown:
for delta_row, delta_col in delta:
next_row = tree[0] + delta_row
next_col = tree[1] + delta_col
if 0 <= next_row < N and 0 <= next_col < N:
trees[(next_row, next_col)] = [1] * grown[tree] + trees[(next_row, next_col)]
total_tree += grown[tree]
for i in range(N):
for j in range(N):
land_energy[i][j] += A[i][j]
print(total_tree)
if __name__ == "__main__":
solution()
10 1 1000
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
100 100 100 100 100 100 100 100 100 100
1 1 1